Stage césure (+2A possible) : télédétection en Europe
avril 12, 2021365FarmNet, CLAAS et John Deere lancent une solution Cloud to Cloud collaborative
avril 13, 2021Il y a de cela deux ans se tenait le 13ème CASP (Critical Assessment of Structure Prediction), une compétition bisannuelle dont le but est de déterminer la structure tri-dimensionnelle d’une sélection de protéines dont la structure actuelle vient d’être élucidée. Il s’agit donc d’une détermination à l’aveugle permettant de suivre les progrès effectués en matière de prédiction de la structure des protéines. En 2018, l’intelligence artificielle Alpha Fold avait remporté la compétition en impressionnant la communauté grâce à un score jamais atteint. Et on apprenait, le 30 Novembre 2020, que la deuxième version d’Alpha Fold avait battu, et de loin, son propre score. Revenons sur cette intelligence qui suscite de grands espoirs dans la communauté biologique internationales.
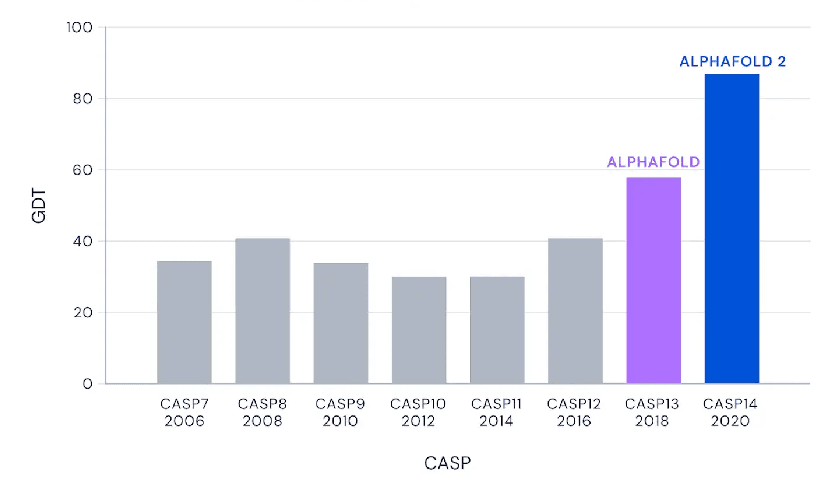
Pour comprendre l’engouement autour d’Alpha Fold, il faut se pencher sur cette notion de score. Le principal élément est ce que l’on appelle le GDT (global distance test), variant de 0 à 100, il représente, pour simplifier, le pourcentage de résidus d’acides aminés situé à une distance seuil de la véritable position.
(source : https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology)
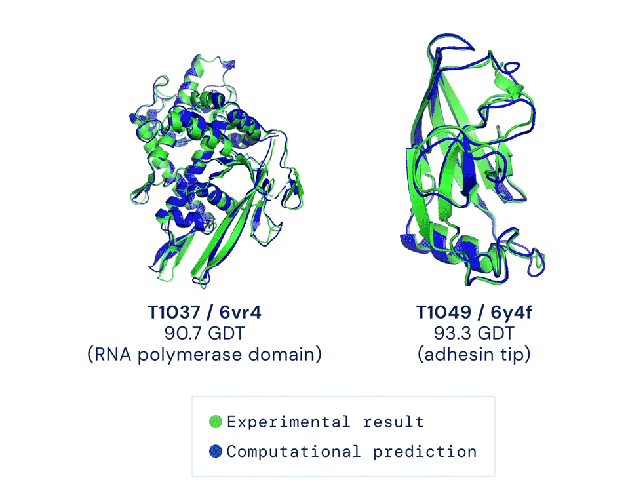
Alpha Fold 1 (celui de 2018) avait dépassé les 50, loin devant les gagnants des éditions précédentes. Mais Alpha Fold 2 (2020) a réussi l’exploit d’atteindre un score médian de 92,4 pour l’ensemble des protéines. Pour bien se représenter ce résultat, il faut préciser qu’à partir de 90 GDT, on considère la méthode comme compétitive avec les méthodes expérimentales.
(source : https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology)
Parlons justement de ces méthodes expérimentales et de leurs limites pour montrer ce que pourrait changer Alpha Fold. Actuellement, ces techniques expérimentales comprennent, par exemple, la cristallographie par rayons X, la résonance magnétique nucléaire ou encore la microscopie cryo-électronique. Si ces techniques permettent de déterminer de manière très précise la structure des protéines, elles sont aussi très onéreuses aussi bien en temps qu’en argent. Elles peuvent prendre des années de travail pour une seule structure et recourent à des équipements de plusieurs millions d’euros. Si bien que sur les 180 millions de séquences protéiques répertoriées dans la base Universal Protein, seules 170 000 ont une structure connue dans la banque de données sur les protéines. Alpha Fold a effectué son apprentissage sur ces 170 000 structures et sur des protéines de structure inconnue en quelques semaines.
Compte tenu de la quantité de structures protéiques inconnues, il n’est pas interdit de rêver lorsque l’on constate le progrès apporté par Alpha Fold 2. Combien de structures possèdent des propriétés inconnues ? Les domaines utilisant les protéines sont trop nombreux pour être tous cités mais vont de l’agroalimentaires, à la gestion des déchets en passant par la médecine. Alpha Fold s’est d’ailleurs illustré récemment dans ce dernier domaine par la détermination de structures protéiques du SRAS-CoV-2.
Il convient cependant de rester mesuré. Les équipes travaillant sur l’IA Alpha Fold n’ont pas encore sorti de papiers pour expliquer concrètement les changements apportés entre AlphaFold1 Et AlphaFold2. De plus, la compétition CASP ne concerne que des domaines de protéines et non des protéines entières. On ne sait pas non plus le temps qu’AlphaFold a pris pour déterminer les structures protéiques du CASP 2020, seulement le temps de son entrainement, même s’il restera beaucoup plus rapide que les techniques expérimentales. Enfin, comme toute intelligence entrainée au deep learning, ses résultats sont très dépendants du jeu d’apprentissage. Il est probable qu’il ne soit pas capable de déterminer la structure d’une protéine atypique.
Sources :
Protein structure prediction beyond AlphaFold, 09 august 2019, Guo-Wei Wei (https://www.nature.com/articles/s42256-019-0086-4)
AlphaFold: a solution to a 50-year-old grand challenge in biology, 30 novembre 2020, the AlphaFold team (https://deepmind.com/blog/article/alphafold-a-solution-to-a-50-year-old-grand-challenge-in-biology)



